Código
library(purrr)
library(dplyr)
library(ggplot2)Pedro de Brito Neto
1 de agosto de 2021
Neste post vamos falar de mais um pacote da família tidyverse: o purrr. O purrr é um pacote que possui diversas ferramentas para trabalhar com funções e vetores. Muitas funções do pacote tem como objetivo aprimorar o uso da programação funcional (FP, do inglês functional programming), já que o R não é uma linguagem diretamente ligada a FP. FP é, em resumo, quando seu código é organizado em funções que realizam as operações de que você precisa, ou seja, em muitos casos você cria suas próprias funções. Aqui vamos tentar mostrar como usar o purrr focando na sua principal função: map().
Como já dito, o pacote pertence ao tidyverse então você pode carregá-lo pelo usando o comando library(tidyverse) ou separadamente library(purrr). Para os exemplos do post iremos trabalhar com um banco de dados iris que o próprio R possui. Você pode carregar bases de dados que o R possui utilizando o comando data()
Dentre as diversas funções do pacote purrr uma das mais conhecidas e utilizadas é a map() e suas variações, nada mais justo do que começar falando dela. Essa função (e suas variações) é usada para aplicar a mesma ação/função a todos elementos de um objeto. Esses objetos podem ser uma lista, um vetor, variáveis de um banco de dados, etc. Esse tipo de função é muito útil para evitar usar loops e for’s.
Se em algum momento você já fez uso da família de funções apply() você pode ter percebido uma certa semelhança entre elas. Existem algumas discussões entre o uso das funções apply() e map(). Não é o foco deste post fazer uma comparação aprofundada sobre essas funções, mas podemos trazer alguns pontos. De forma geral, escolher qual delas usar vai depender de um contexto. Em questão de tempo bruto de execução, a família apply() é ligeiramente mais rápida (a diferença é bem pequena), porém as funções de map() são mais convenientes. Todas as suas variações seguem a mesma ordem de argumentos (o primeiro argumento é sempre o vetor), diferente da família apply(), onde os argumentos podem mudar. Existem mais pontos a serem considerados, mas em resumo, se ignorar questões de sintaxe e funcionalidade (as variações de map()), decidir qual delas usar restringe-se a usar as funções base do R ou utilizar funções obtidas por meio de pacotes, neste caso o purrr.
Voltando ao foco, vamos mostrar algumas variações (não todas) de map().
map(.x, .f) é a principal função de mapeamento e retorna uma lista.
map_df(.x, .f) retorna um data.frame.
map_dbl(.x, .f) retorna um vetor numérico (duplo).
map_chr(.x, .f) retorna um vetor de caracteres.
map_lgl(.x, .f) retorna um vetor lógico.
Essas funções, assim como a maiores das funções do universo tidyverse, tem como primeiro argumento o objeto a qual você deseja aplicar uma certa função e o segundo argumento é a função a ser aplicada em cada elemento do seu objeto. Para ficar mais claro vamos a uma breve aplicação e ao decorrer do post, iremos utilizar mais as outras funções. Vamos criar um vetor com poucos elementos para usar como exemplo, depois criar uma função qualquer e usar o map() para aplicar essa função em cada elemento do vetor.
| [[1]] | |
| [1] | 0 |
| [[2]] | |
| [1] | 9 |
| [[3]] | |
| [1] | 36 |
| [[4]] | |
| [1] | 81 |
| [[5]] | |
| [1] | 144 |
| [1] | “0.000000” | “9.000000” | “36.000000” | “81.000000” | “144.000000” |
Note que usar apenas map(), como dito antes, irá retornar uma lista onde o primeiro elemento da saída é o resultado da aplicação da função ao primeiro elemento da entrada e assim por diante. Já quando usamos map_chr() a função nos retorna um vetor de caracteres.
Vamos agora criar uma lista com alguns vetores de tamanhos diferentes. Caso a gente queira, por exemplo, tirar a média de cada um dos vetores dentro da lista, precisaríamos fazer isso acessando cada elemento da lista, criando funções complexas ou utilizando for’s e loops. Veja abaixo como o map() pode nos ajudar.
| [[1]] | |||||||||||
| [1] | 1 | 2 | 3 | ||||||||
| [[2]] | |||||||||||
| [1] | 0 | 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 |
| [[3]] | |||||||||||
| [1] | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 |
| [[1]] | |
| [1] | 2 |
| [[2]] | |
| [1] | 50 |
| [[3]] | |
| [1] | 5.5 |
| [1] | 1.00000 | 33.16625 | 3.02765 |
Note também que fizemos o uso da função junto com o pipe (%>%) e é bem comum essa prática. Lembrando que já explicamos como utilizar o pipe, caso tenha alguma dúvida você pode acessar o post que contém essa explicação clicando aqui.
Nos exemplos acima utilizamos uma função criada por nós e outras já presentes dentro do R. Além dessas, também é possível utilizar o que chamamos de funções anônimas. Uma função anônima é basicamente uma função criada e usada, mas nunca atribuída a um objeto. Esse tipo de função é bem útil dentro do map(). Para usar essa função você começa com um ~ (para indicar que você começou uma função anônima). O argumento da função é consultado utilizando o .x ou apenas ., diferente das funções normais onde você consegue usar qualquer argumento. Para ficar mais claro vamos a um exemplo.
| [1] | 0 | 9 | 36 | 81 | 144 |
| [1] | 0 | 9 | 36 | 81 | 144 |
| [1] | 0 | 9 | 36 | 81 | 144 |
| [1] | 0 | 9 | 36 | 81 | 144 |
Acima mostramos maneiras diferentes de utilizar funções anônimas. Todas retornam o mesmo resultado (e deveriam). Um destaque para a última onde foi usado {}. O uso das chaves é interessante para organização e boas práticas no seu código, principalmente se a sua função for mais complexa.
Uma função muito útil também é a modify(). Ela funciona de forma igual à função map(), a diferença é que ela sempre irá retornar um objeto do mesmo tipo que o objeto de entrada. Por exemplo, se meu objeto de entrada for um vetor de caracteres, a saída também será um vetor de caracteres. Note que isso pode ser considerado como uma boa prática e já fizemos um post sobre isso no software R disponível aqui.
| x | y | |
| 1 | 1 | 81 |
| 2 | 100 | 400 |
| 3 | 121 | 900 |
A função modify() possui uma variação interessante. A modify_if() permite que você forneça um critério e assim, a função só será aplicada aos elementos que atendam a esse critério. O critério torna-se o segundo argumento da função, ou seja, o primeiro argumento continua sendo o objeto de entrada, o segundo passa a ser o critério e o terceiro argumento é a função. Para ficar mais claro vamos a um exemplo
| [1] | 0 | 3 | 6 | 81 | 144 |
Veja que a função só foi aplicada para valores maiores ou iguais a 9.
Agora que temos uma noção de como funciona as funções map(), vamos deixar as coisas um pouco mais interessantes trabalhando com uma base de dados. Para os exemplos do post iremos trabalhar com um banco de dados “iris” que o próprio R possui. Você pode olhar bases de dados que o R possui utilizando o comando data()
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species |
|---|---|---|---|---|
| 5.1 | 3.5 | 1.4 | 0.2 | setosa |
| 4.9 | 3.0 | 1.4 | 0.2 | setosa |
| 4.7 | 3.2 | 1.3 | 0.2 | setosa |
| 4.6 | 3.1 | 1.5 | 0.2 | setosa |
| 5.0 | 3.6 | 1.4 | 0.2 | setosa |
| 5.4 | 3.9 | 1.7 | 0.4 | setosa |
| 4.6 | 3.4 | 1.4 | 0.3 | setosa |
| 5.0 | 3.4 | 1.5 | 0.2 | setosa |
| 4.4 | 2.9 | 1.4 | 0.2 | setosa |
| 4.9 | 3.1 | 1.5 | 0.1 | setosa |
Como estamos trabalhando com uma base de dados, as funções serão aplicadas em cada coluna. Por exemplo, ao aplicar uma função para resumir de alguma maneira os dados, ela irá fazer isso para cada uma das colunas. Vamos começar utilizando a função n_distinct() do pacote dplyr que nos retorna o número de valores distintos em cada coluna.
$Sepal.Length |
|
| [1] | 35 |
$Sepal.Length |
|
| [1] | 23 |
$Petal.Length |
|
| [1] | 43 |
$Petal.Length |
|
| [1] | 22 |
$Species |
|
| [1] | 3 |
Observe que map() aplicou a função desejada em cada uma das 5 variáveis do nosso banco de dados e retornou o valor correspondente.
Vamos começar a fazer algo mais elaborado. Podemos querer retornar algumas medidas resumos ou outras informações gerais sobre nossos dados em uma base de dados. Esse é um momento bem legal e útil de utilizar o map_df(), uma das funções mais poderosas do pacote. Basicamente utilizamos a estrutura map_df( ~ data_frame(x = .x" ), onde x = o nome da coluna e .x a função anônima. Pra ficar mais claro, vamos à pratica.
No exemplo acima, começamos utilizando a função select_if() do pacote dplyr para pegar do banco de dados original apenas as variáveis do tipo númericas e armazenamos em um outro banco de dados (apenas para facilitar o entendimento). Após isso já começamos a pensar na medidas que queremos para acrescentar no banco de dados. Adicionamos então os valores distintos de cada variável, as classes (apenas para mostrar que select_if() fez o que queriamos) e algumas medidas resumo como média, desvio padrão, valores máximos e mínimos. Uma observação importante é que ao utilizar essa função de map(), o nome das variáveis são perdidos, por isso ao fim do código foi utilizado o .id (argumento de map_df()) para incluir o nome das variáveis. Veja o resultado.
| variavel | v_distintos | classe | media | desvio_p | minimo | maximo |
|---|---|---|---|---|---|---|
| Sepal.Length | 35 | numeric | 5.84 | 0.83 | 4.3 | 7.9 |
| Sepal.Width | 23 | numeric | 3.06 | 0.44 | 2.0 | 4.4 |
| Petal.Length | 43 | numeric | 3.76 | 1.77 | 1.0 | 6.9 |
| Petal.Width | 22 | numeric | 1.20 | 0.76 | 0.1 | 2.5 |
Após obtermos uma certa noção de como funcionam as funções de map() podemos falar de sua outra versão, a map2(). Essa função segue os mesmos critérios que a map() “original”, porém permite que você trabalhe não só com um, mas com dois objetos. Em map2() também conseguimos especificar o tipo de saída da função, segue abaixo a lista de algumas das variações da função.
map2 (.x, .y, .f, ...)
map2_dfc (.x, .y, .f, ...)
map2_dbl (.x, .y, .f, ...)
map2_chr (.x, .y, .f, ...)
map2_lgl (.x, .y, .f, ...)
Lembrando que .x e .y são os vetores da função e devem ter o mesmo tamanho, caso contrário a função retornará uma mensagem de erro.
summarise() has grouped output by ‘Sepal.Width’. You can override using the .groups argument. |
| [[1]] | |
| [1] | “Espécie: versicolor largura: 2” |
| [[2]] | |
| [1] | “Espécie: versicolor largura: 2.2” |
| [[3]] | |
| [1] | “Espécie: virginica largura: 2.2” |
| [[4]] | |
| [1] | “Espécie: setosa largura: 2.3” |
| [[5]] | |
| [1] | “Espécie: versicolor largura: 2.3” |
Acima criamos dois vetores, cada um contendo uma variável do banco de dados e depois criamos uma função simples, que irá concatenar, nesse caso, os dois vetores com algumas frases. Note que a função foi aplicada aos dois vetores. Uma observação é que no começo do exemplo criamos um outro banco de dados (dados3) com apenas as duas variáveis que foi usada no exemplo e agrupamos elas utlizando group_by() esummarise(). Isso foi feito apenas para que fosse possível ver os resultados com observações diferentes na saida da função.
É possível utilizar o map() para mais de dois objetos. Neste caso não existe um map3() ou map4() mas sim o pmap(). pmap() é basicamente uma generalização de map2() para 3 ou mais objetos. Essa função possui uma pequena diferença, nesse caso você não irá especificar n objetos dentro da função, mas sim apenas uma única lista que contém todos os vetores (ou listas).
Para esse exemplo, vejamos a função split que nos será útil. Usaremos essa função que divide os dados de entrada (x) em grupos diferentes (f). Um breve exemplo de como a função funciona.
| a | b | b | a | a |
| “1” | “25” | “exemplo” | “split” | “3” |
$a |
|||
| a | a | a | |
| “1” | “split” | “3” | |
$b |
|||
| b | b | ||
| “25” | “exemplo” |
Note que o objeto sp foi separado em duas listas de acordo com os nomes do vetor.
Agora considerando os dados iris, separamos os dados em listas de acordo com as espécies. No caso do nosso exemplo o objeto “especies” vai ser uma lista com as 3 especies presentes no banco de dados iris.
Uma forma de observar o resultado obtido é verificar a classe e as dimensões de cada uma das partes obtidas, utilizando a função map.
| setosa | versicolor | virginica |
| “data.frame” | “data.frame” | “data.frame” |
$setosa |
||
| [1] | 50 | 5 |
$versicolor |
||
| [1] | 50 | 5 |
$virginica |
||
| [1] | 50 | 5 |
Para checar que a divisão foi feita de forma correta, podemos avaliar o banco de dados original e contar a quantidade de observações para cada tipo de espécie.
| setosa | versicolor | virginica |
| 50 | 50 | 50 |
Depois partimos para criar o nosso modelo. Com ajuda da função map(), utilizamos a função lm() para ajustar um modelo de regressão linear. A função lm() possui vários argumentos e maneiras de utilizar, não é nosso foco aprofundar nessa função, mas você pode saber mais clicando aqui. De maneira geral, fornecemos à função uma variável preditora (ou independente) que nesse caso foi Petal.Length e a variável dependente (aquela que estamos tentando explicar) que foi Sepal.Length. O segundo argumento é o banco de dados, nesse caso utilizamos ., pois gostaríamos de ajustar um modelo linear para cada elemento da lista de banco de dados. Vamos imprimir o resultado para entender melhor o que foi feito.
$setosa |
| Call: |
| lm(formula = Sepal.Length ~ Petal.Length, data = .) |
| Coefficients: |
| (Intercept) Petal.Length |
| 4.2132 0.5423 |
$versicolor |
| Call: |
| lm(formula = Sepal.Length ~ Petal.Length, data = .) |
| Coefficients: |
| (Intercept) Petal.Length |
| 2.4075 0.8283 |
$virginica |
| Call: |
| lm(formula = Sepal.Length ~ Petal.Length, data = .) |
| Coefficients: |
| (Intercept) Petal.Length |
| 1.0597 0.9957 |
Note que a função estimou uma reta com dois parâmetros: intercepto e a inclinação da reta segundo a variável preditora. Essa reta tenta explicar o comprimento médio da pétala (Petal.Length) como função do comprimento da sépala (Sepal.Length). Repare que os interceptos são bem distintos entre os três ajustes e a inclinação entre as espécies versicolor e virginica são bem parecidas, de acordo com o resultado impresso. Esse resultado pode ser visualizado com a ajuda do pacote ggplot2 com o seguinte código.
geom_smooth() using formula ‘y ~ x’ |
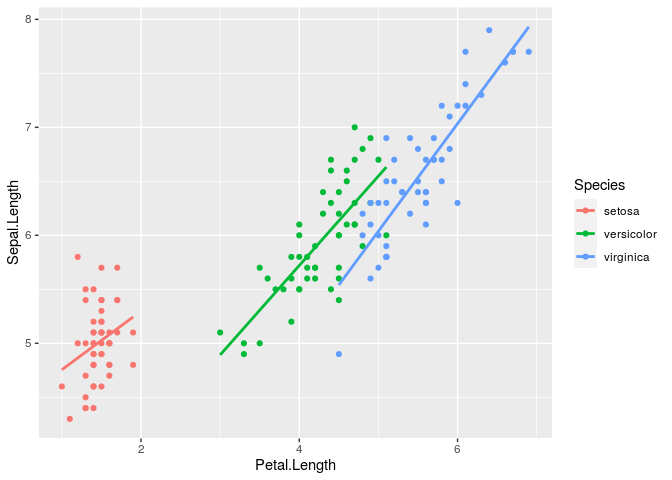
Note que as retas estimam uma relação linear entre o valor médio do comprimento da pétala como função do comprimento da sépala, para cada uma das espécies.
Por fim fizemos o uso de map2() com os os objetos “modelos” e “especies”, e aplicamos a função predict(). Em resumo, predict() serve para prever valores tendo como base um modelo (você pode se aprofundar nessa função clicando aqui). Nesse caso, estamos obtendo as predições de cada um dos modelos estimados, para cada uma das observações considerando os diferentes bancos de dados, armazenados no objeto especies. Podemos visualizar os resultados obtidos fazendo um gráfico simples de pontos e comparar com o gráfico anterior do modelo estimado.
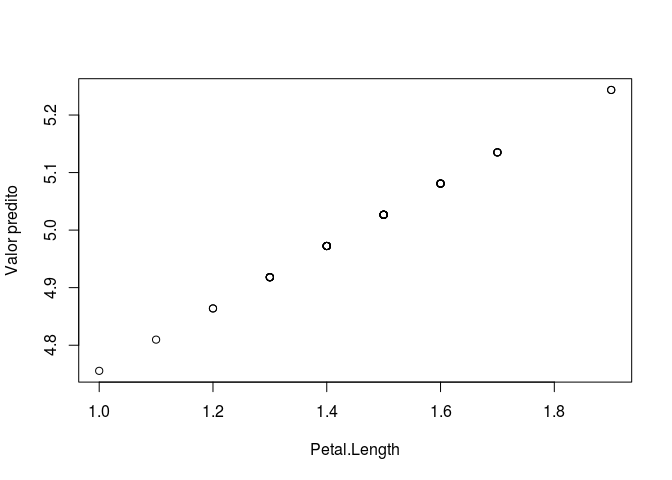
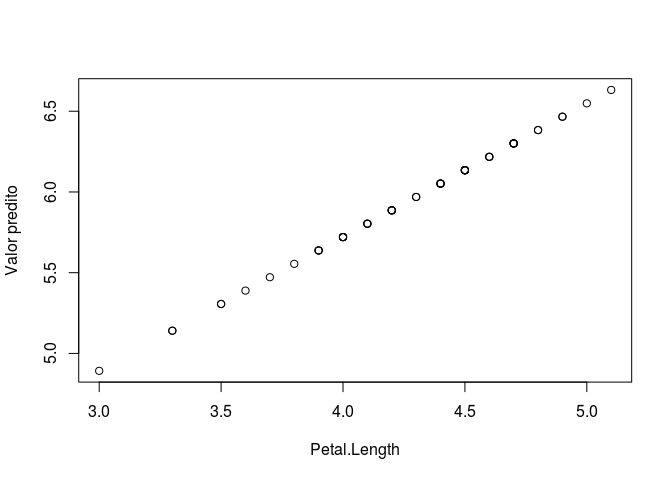
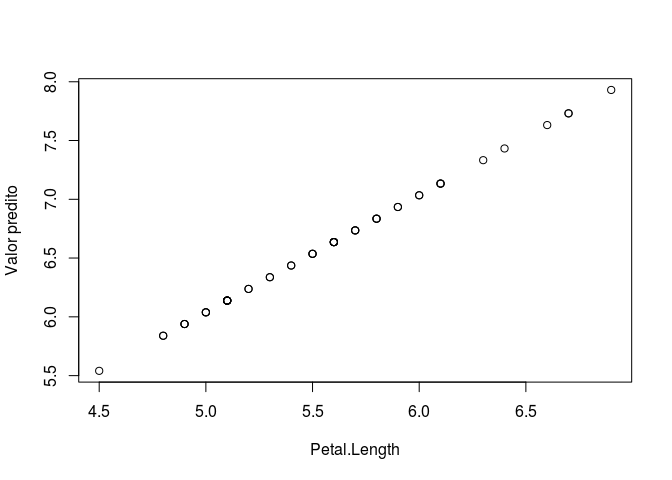
Nesse exemplo, nós utilizamos o comprimento do objeto predicoes para acessar as diferentes predições e suas respectivas variáveis preditoras. Veja que como os dois objetos especies e predicoes são uma lista, nós utilizamos o operador [[]] para acessar suas posições. Note que para cada valor de x (Petal.Length) existe um valor predito segundo o modelo ajustado e todos estão descritos de acordo com uma reta, que foi aquela estimada segundo a função lm. A função invisible() foi utilizada somente para evitar a impressão de uma lista de valores NULL ao final do processo. Veja como seria o resultado sem essa função e compare.
Para mostrar algumas funções do purrr que nos auxiliam na manipulação de listas, vamos transformar o banco de dados que estamos usando (iris) em uma lista de banco de dados. Já fizemos isso no exemplo anterior, mas aqui para garantir que seja fácil de seguir e visualizar, vamos usar apenas as primeiras 5 linhas de cada espécie.
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species | |
| 1 | 5.1 | 3.5 | 1.4 | 0.2 | setosa |
| 2 | 4.9 | 3.0 | 1.4 | 0.2 | setosa |
| 3 | 4.7 | 3.2 | 1.3 | 0.2 | setosa |
| 4 | 4.6 | 3.1 | 1.5 | 0.2 | setosa |
| 5 | 5.0 | 3.6 | 1.4 | 0.2 | setosa |
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species | |
| 1 | 7.0 | 3.2 | 4.7 | 1.4 | versicolor |
| 2 | 6.4 | 3.2 | 4.5 | 1.5 | versicolor |
| 3 | 6.9 | 3.1 | 4.9 | 1.5 | versicolor |
| 4 | 5.5 | 2.3 | 4.0 | 1.3 | versicolor |
| 5 | 6.5 | 2.8 | 4.6 | 1.5 | versicolor |
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species | |
| 1 | 6.3 | 3.3 | 6.0 | 2.5 | virginica |
| 2 | 5.8 | 2.7 | 5.1 | 1.9 | virginica |
| 3 | 7.1 | 3.0 | 5.9 | 2.1 | virginica |
| 4 | 6.3 | 2.9 | 5.6 | 1.8 | virginica |
| 5 | 6.5 | 3.0 | 5.8 | 2.2 | virginica |
A função keep() e discard() são bem parecidas e opostas. keep() mantém apenas os elementos de uma lista que satisfazem uma determinada condição, enquanto discard() faz exatamente o oposto (descarta quaisquer elementos que satisfaçam a condição lógica). Essas funções possuem 2 argumentos principais, .x uma lista ou vetor e .p uma função. Apenas aqueles elementos .p avaliados como TRUE serão mantidos ou descartados. Também é possível, como terceiro argumento, fornecer argumentos adicionais transmitidos para “.p”. A função compact() tem argumentos parecidos com as funções anteriores, a diferença é que para essa função, o .p é uma função que é aplicada a cada elemento de .x e somente aqueles elementos .p avaliados como um vetor vazio (NULL ou listas com comprimento zero) serão descartados.
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species | |
| 1 | 7.0 | 3.2 | 4.7 | 1.4 | versicolor |
| 2 | 6.4 | 3.2 | 4.5 | 1.5 | versicolor |
| 3 | 6.9 | 3.1 | 4.9 | 1.5 | versicolor |
| 4 | 5.5 | 2.3 | 4.0 | 1.3 | versicolor |
| 5 | 6.5 | 2.8 | 4.6 | 1.5 | versicolor |
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species | |
| 1 | 6.3 | 3.3 | 6.0 | 2.5 | virginica |
| 2 | 5.8 | 2.7 | 5.1 | 1.9 | virginica |
| 3 | 7.1 | 3.0 | 5.9 | 2.1 | virginica |
| 4 | 6.3 | 2.9 | 5.6 | 1.8 | virginica |
| 5 | 6.5 | 3.0 | 5.8 | 2.2 | virginica |
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species | |
| 1 | 5.1 | 3.5 | 1.4 | 0.2 | setosa |
| 2 | 4.9 | 3.0 | 1.4 | 0.2 | setosa |
| 3 | 4.7 | 3.2 | 1.3 | 0.2 | setosa |
| 4 | 4.6 | 3.1 | 1.5 | 0.2 | setosa |
| 5 | 5.0 | 3.6 | 1.4 | 0.2 | setosa |
$a |
|
| [1] | “a” |
$d |
|
| [1] | NA |
Caso a gente queira saber se todos, alguns ou nenhum dos elementos de uma lista satisfazem um determinado critério, podemos usar as funções every(), some() e none(), respectivamente. Essas funções são básicas e possuem apenas dois argumentos, sendo o primeiro uma lista ou vetor, e o segundo argumento é o critério. A saída da função será valores lógicos (TRUE ou FALSE).
| [1] | FALSE |
| [1] | TRUE |
| [1] | TRUE |
Aqui podemos puxar um gancho para uma outra fução de pesquisa que também retorna um valor lógico, has_element. Basicamente serve para testar se uma lista ou vetor contém um determinado objeto, a função tem o seguinte formato:
has_element(.x, .y)onde .x é a lista ou vetor e .y o objeto para testar
| [1] | TRUE |
| [1] | FALSE |
A função detect() serve para encontrar o valor ou posição da primeira correspondência. Em outras palavras, ela retorna o primeiro elemento que passa no teste ou condição lógica.
detect(.x, .f, ..., .right = FALSE, .p).x é o argumento da função onde você fornece uma lista ou vetor atômico, .f uma função, fórmula ou vetor atômico.
Se uma fórmula , por exemplo ~ .x + 2, ela é convertida em uma função. Existem três maneiras de se referir aos argumentos:
Para uma função de argumento único, use .
Para uma função de dois argumentos, use .x e .y
Para mais argumentos, utilização ..1, ..2, ..3 etc
Vamos a um exemplo simples para entender melhor a função.
| [1] | 4 |
Como dito antes, ela irá retornar o primeiro valor que responde ao critério usado (segundo argumento). Quando se trata de funções deste tipo (detectar), o pacote stringr pode ser mais interessante. Já fizemos um post muito massa sobre este pacote, basta clicar aqui para acessá-lo.
O purrr possui diversas funções, muitas delas não aparecem nesse post. Aqui focamos em explicar as funções que são mais utilizadas e algumas mais simples. As funções de purrr possuem uma sintaxe muito parecida, então se entender bem as funções que trouxemos aqui, as outras serão bem mais fáceis de entender. Vale lembrar que você pode acessar o cheatsheet para ver todas as funções clicando aqui.
https://www.rebeccabarter.com/blog/2019-08-19_purrr/
https://jennybc.github.io/purrr-tutorial/
https://www.r-bloggers.com/2020/05/one-stop-tutorial-on-purrr-package-in-r/