Mergulhando no tidyverse
Na maioria dos trabalhos e análises que vamos fazer aqui no R, sempre
precisamos de pacotes que nos auxiliem, seja um que facilite o
tratamento de dados ou um que apenas deixa a estétíca de um gráfico um
pouquinho mais legal. De qualquer forma, sempre precisamos carregar
pacotes para o nosso projeto. Nesse post vamos falar um pouco sobre um
dos pacotes mais utilizados no R, o tidyverse. O tidyverse é uma
coleção de outros pacotes que são utilizados para manipulação,
exploração e visualização de dados e que compartilham uma filosofia de
design bem parecida, por isso de forma combinada permitem que você
consiga fazer inúmeros trabalhos. Os pacotes que fazem parte desse
universo são: dplyr, tidyr, ggplot2, forcats, purrr,
stringr, tibble e readr. Para descrever melhor como funciona o
tidyverse, vamos falar sobre cada pacote presente nele de uma forma
bem resumida, mostrando suas principais funções e exemplos de como
utilizar. Mais para frente criaremos também posts para falar desses
pacotes de uma forma mais aprofundada.
Para mostrar a utilização do tidyverse iremos realizar um tratamento
na base de dados do censo demográfico brasileiro, em que o último foi
realizado em 2010. Iremos também usar uma base de dados que contém
informações da população estimada em cada município do Brasil em 2020.
Com a utilização de algumas funções do pacote será possível juntar os
dois bancos de dados e realizar uma comparação da população.
#carregando o tidiverse
library(tidyverse)
readr
O readr é um pacote que serve para ler e importar bancos de dados para
o R, os dados podem ser de diversos tipo, como, csv, tsv e xlsx. O
readr combina duas partes para fazerem as funções, onde uma irá
analisar o arquivo de maneira geral e outra a especificação da coluna. A
especificação da coluna descreve como cada coluna deve ser convertida de
um vetor de caracteres para o tipo de dados mais apropriado e, na
maioria dos casos, não é necessário porque readr irá fazê-lo
automaticamente. Já fizemos um post sobre importação de dados no R
onde mostramos diferentes maneiras de importar dados, inclusive usando o
readr. Você pode acessar o post clicando
aqui.
read_csv(): arquivos separados por vírgula (CSV)read_tsv(): arquivos separados por tabulaçãoread_delim(): arquivos gerais delimitadosread_fwf(): arquivos de largura fixaread_table(): arquivos tabulares onde as colunas são separadas por-
- espaço em branco.
-
read_log(): arquivos de log da web
Para as análises, estou importando os dados com a função read_excel do
pacote readxl. Para reprodução do post, os arquivos podem ser obtidos
aqui e aqui.
library(readxl)
censo <- read_excel("censo.xlsx")
estimado <- read_excel("estimado.xlsx")
Após a importação dos dados, iremos começar o tratamento, para isso
iremos usar os pacotes dplyr, forcats e algumas funções já
existentes no pacote básico do R. Então, vamos primeiro explicar de
forma rápida o que cada um desses pacotes pode fazer.
dplyr
Primeiramento vamos falar sobre o pacote dplyr. O pacote tem uma
descrição bem direta para mostrar pra que ele serve: “dplyr é uma
gramática de manipulação de dados, fornecendo um conjunto consistente
de verbos que ajudam a resolver os desafios mais comuns de manipulação
de dados”. Para maiores informações acesse este
link Vamos então mostrar
algumas de suas funções.
mutate(): adiciona novas variáveis que são funções de variáveis existentes.group_by(): agrupa pela(s) variável(is) no argumento. Função muito útil quando usada a funçaõ summurise.summarise(): reduz vários valores a um único resumo.select(): escolhe variáveis com base em seus nomes.filter(): escolhe casos com base em seus valores.arrange(): altera a ordem das linhas.
Essas funções são bastante utilizadas em diversos tipos de análises e cada uma com uma finalidade, podemos misturar essas funções entre si e até mesmo com funções de outros pacotes para gerar o resultado desejado.
O dplyr também carrega uma função extremamente utilizada no R, o pipe
(%>%). Ele é usado para expressar claramente uma sequência de
múltiplas operações. Mas porque devo usar o pipe? Bom, veja algumas
vantagens:
-
Você estruturará a sequência de suas operações de dados da esquerda para a direita, em oposição à sequência de dentro para fora;
-
Você evitará chamadas de função aninhadas;
-
Você minimizará a necessidade de variáveis locais e definições de funções;
-
Você tornará mais fácil adicionar etapas em qualquer lugar na sequência de operações.
Voltando para as análises. Primeramente vamos arrumar a base de dados do
censo, usaremos a função clean_names do pacote janitor (não
pertencente ao tidyverse) para limpar e padronizar os nomes das
variáveis, depois aplicaremos um filtro para pegar apenas os dados de
- Iremos renomear a varíavel
ufparauf_codepara facilitar um pouco nosso entendimento. Note que já estamos usando algumas das funções mencionadas.
censo <- janitor::clean_names(censo)
censo <- censo %>%
filter(ano == 2010) %>%
mutate(uf = as.character(uf)) %>%
rename("uf_codigo" = uf)
forcats
O forcats é um pacote com diversas funções com o objetivo de resolver problemas com fatores (variáveis categóricas). Por exemplo, esse pacote pode ser usado para alteração da ordem dos níveis (categorias) ou dos valores dos fatores. Vamos ver algumas de suas principais funções:
fct_reorder(): reordenando um fator por outra variável.fct_infreq(): reordenando um fator pela frequência dos valores.fct_relevel(): alterar a ordem de um fator manualmente.fct_lump(): colapso dos valores menos / mais frequentes de um fator em “outro”.fct_recode(): permite que você recodifique ou altere o valor de cada nível.
Agora iremos criar uma nova variável com a função mutate que irá
receber os nomes dos municípios, porém todos em letras minúsculas com o
auxilio da função tolower. Em seguida, criaremos mais uma variável que
vai pegar os códigos dos municípios presentes na vaiável uf_code e
recodificá-los utilizando a função fct_recode do forcats para o
estado a qual se refere. Os códigos dos municípios foram obtidos no site
do IBGE.
censo <- censo %>%
mutate(municipio_1 = tolower(municipio))
censo <- censo %>%
mutate(uf = fct_recode(uf_codigo,
"RO" = "11",
"AC" = "12",
"AM" = "13",
"RR" = "14",
"PA" = "15",
"AP" = "16",
"TO" = "17",
"MA" = "21",
"PI" = "22",
"CE" = "23",
"RN" = "24",
"PB" = "25",
"PE" = "26",
"AL" = "27",
"SE" = "28",
"BA" = "29",
"MG" = "31",
"ES" = "32",
"RJ" = "33",
"SP" = "35",
"PR" = "41",
"SC" = "42",
"RS" = "43",
"MS" = "50",
"MT" = "51",
"GO" = "52",
"DF" = "53"
))
censo <- censo %>%
select(ano, uf, municipio_1, municipio, everything()) #essa função apenas reordena as variáveis.
Vamos agora tratar os dados da segunda base de dados, que contém os
dados da população estimada. Aqui fizemos tudo de uma maneira mais
direta utilizando o pipe. Agora iremos utilizar algumas funções do
pacote stringr então, vamos falar um pouco sobre ele.
stringr
O pacote stringr possui diversas funções para nos ajudar no tratamento
de strings. Mas afinal, o que é uma string? Em resumo, uma string é umas
sequência de caracteres que geralmente são utilizadas para representar
palavras, frases ou textos de um programa. Strings estão presentes da
maioria dos bancos de dados e é importante ter pelo menos noção de como
“organizar” essas strings. Seguem algumas funções:
str_detect(): informa se há alguma correspondência com o padrão.str_count(): conta o número de padrões.str_subset(): extrai os componentes correspondentes.str_locate(): dá a posição da partida.str_extract(): extrai o texto da partida.str_replace(): substitui as correspondências por um novo texto.str_split(): divide uma string em vários pedaços.
Para aprofundar melhor no uso dessas funções, iremos deixar o
link
do cheat sheet (folha de dicas) do stringr.
Agora que já entendemos um pouco o funcionamento do stringr, podemos
usar suas funções para auxiliar nossa análise. A variável municipio
desse banco de dados vem no formato “nome do município (uf)” logo, é
necessário utilizar uma função que “entra” em cada observação dessa
variável e pegue apenas o “UF” (estado). Depois disso criaremos outra
variável que irá remover o “UF” e fique apenas o nome do município.
Utilizamos a função str_match e str_sub, respectivamente.
estimado <- janitor::clean_names(estimado)
estimado <- estimado %>%
mutate(uf = str_match(municipio, "[(]([^)]+)[)]")[,2]) %>% #essa função irá pegar as palavras dentro de parêntes, ex: "(MG)"
mutate(muni = str_sub(municipio, end = -6)) %>% # aqui estamos removendo os últimos dígitos (UF´s) e ficando apenas com o nome do município
mutate(municipio_1 = tolower(muni)) %>%
select(uf, municipio_1, populacao_estimada, everything())
Agora que tratamos as variáveis desejadas das duas tabelas, é
interessante juntá-las em apenas uma base de dados. Vamos fazer isso
usando uma função presente no pacote dplyr.
dados <- left_join(estimado, censo, by = c("municipio_1", "uf"))
Algumas observações devem ser feitas. Os bancos de dados possuem quantidades diferentes de observações.
nrow(censo) # quantidade de observações do banco de dados do censo
## [1] 5565
nrow(estimado) # # quantidade de observações do banco de dados estimado
## [1] 5571
Como o motivo dessa diferença na quantidade de observações não está
muito claro, optamos em útilizar o left_join que irá retonar todas as
linhas do primeiro banco de dados escolhido, neste caso, pegamos o com o
maior número de observações, e irá atribuir valores vazios para células
não correspondentes dentre os dois bancos de dados.
tidyr
O pacote tidyr foi criado para ajudá-lo a criar dados organizados. Mas o que se caracteriza como dados organizados? Bom, podemos listar algumas considerações como:
- cada coluna é uma variável.
- Cada linha é uma observação.
- Cada célula é um único valor.
Organizar seus dados dessa maneira facilita o trabalho porque você tem
uma maneira consistente de se referir a variáveis (como nomes de coluna)
e observações (como índices de linha). Ao usar dados organizados e
ferramentas organizadas, você perde menos tempo se preocupando em como
alimentar a saída de uma função na entrada de outra e mais tempo
respondendo às suas perguntas sobre os dados. O tidyr descreve o
formato dos dados como long (longo), ou seja, com mais linhas e wide
(amplo), com mais colunas. O pacote possui qutro principais funções para
organizar os seus dados: gather(), separate(), spread() e
unite().
gather(): torna os dados “amplos” mais longos.spread(): torna os dados “longos” mais amplos.separate(): divide uma única coluna em várias colunas.unite(): combina várias colunas em uma única coluna.
Você pode aprender mais sobre essas funções neste
link. O tidyr também possui diversas
outras funções que você pode olhar acessando o
cheatsheets.
Nesta análise iremos utilizar a função drop_na (retirar linhas
contendo valores ausentes) do pacote para remover as variáveis com
valores vazios e selecionar apenas as que iremos utilizar.
dados <- drop_na(dados)
dados <- dados %>%
select(uf, municipio_1, populacao_estimada, pop)
Agora que retiramos os valores vazios que a princípio seriam apenas os municípios não existentes no banco de dados do censo, notamos que a tabela criada possui um número de linhas um pouco menor que a tabela original. Isso se deve ao fato de que alguns municípios foram escritos de formas diferentes em cada tabela, talvez acentuações diferentes ou algum erro de digitação. Como o foco desse post não é fazer uma análise aprofundada dos dados, vamos relevar essa diferença e continuar a análise.
A maneira mais interessantes de podermos comparar essa diferença entre a
população em 2010 e a população estimada em 2020 é por meio de gráficos,
o pacote ggplot2que faz parte do universo tidyverse é um dos mais
utilizados quando o assunto é fazer gráficos. Então, vamos apresentá-lo.
ggplot2
O ggplot é um pacote extremamente utilizado no R que tem como finalidade
criar gráficos de diversos tipos. Basicamente você fornece o conjunto de
dados, diz qual tipo de gráficos utilizar e mostra como mapear as
variáveis para a estética do gráfico. O ggplot2 trabalha com
“camadas” onde, após você atribuir algo ao gráfico, você utiliza o
+ e acrescenta uma nova camada. Para explicar isso de uma forma mais
clara, vamos a um exemplo. Começaremos com ggplot() e nele vamos
inserir o conjunto de dados, depois a camada fornecendo o tipo de
gráfico com os atributos estéticos, por exemplo, geom_bar(aes()). Com
esses comandos já é possível exibir um gráfico, mas podemos colocar
inúmeras camadas.
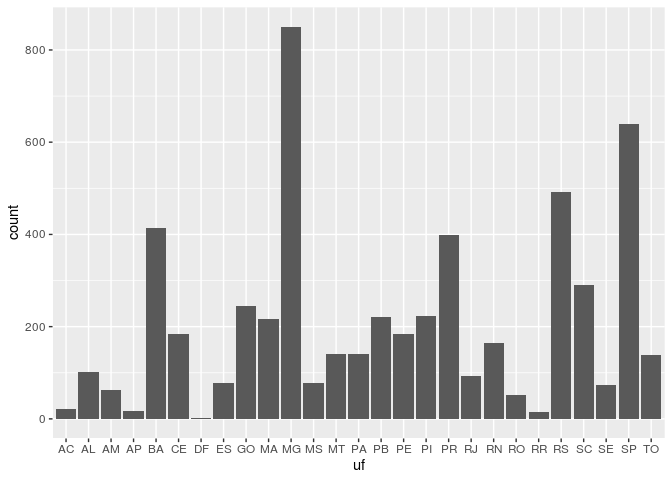
# um exemplo extramente simples que mostra a quantidade de municípios em cada estado.
ggplot(dados) + geom_bar(aes(x = uf))
# podemos aprimorar esse mesmo gráfico
ggplot(dados, aes(x = uf)) + geom_bar(fill = "purple") +
theme_bw() +
scale_y_continuous(breaks = seq(0, 1000, 100)) +
labs(title = "Quantidade de municípios por Estado",
x = "Estado",
y = "Quantidade")
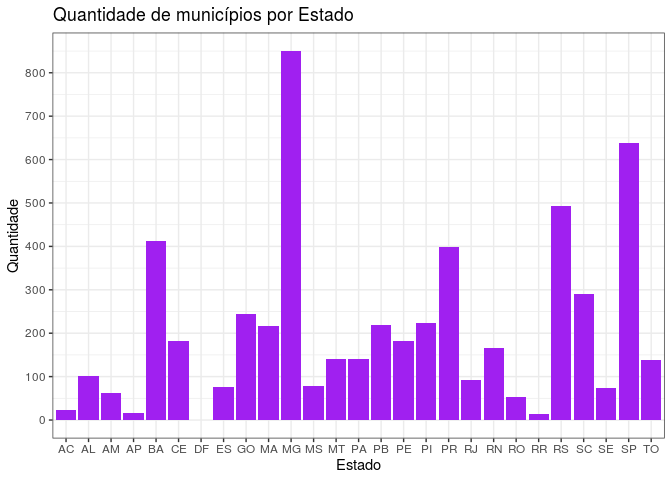
Note que coloquei as informações estéticas com a função aes em lugares
diferentes aqui nesse exemplo e o resultado seria exatamente o mesmo,
porém isso nem sempre é verdade. Em um post somente sobre ggplot2
iremos discutir isso com mais detalhes.
Vamos então aos gráficos de comparação. Para isso, precisaremos plotar
duas variavéis no eixo x, população e população estimada, então
precisaremos fazer uma breve manipulação nos dados. Neste caso, vamos
usar a função melt do pacote reshape2 que irá basicamente juntar em
apenas uma variável: a população e a população estimada.
library(reshape2)
dados_1 <- melt(dados)
ggplot(dados_1, aes(uf, value, fill = variable)) +
geom_bar(stat = "identity", position = "dodge") +
labs(title = "população total em 2010 e população estimada em 2020",
x = "Estado",
y = "Quantidade") +
guides(fill = guide_legend(title = "Legenda")) +
theme_bw() +
scale_fill_viridis_d()
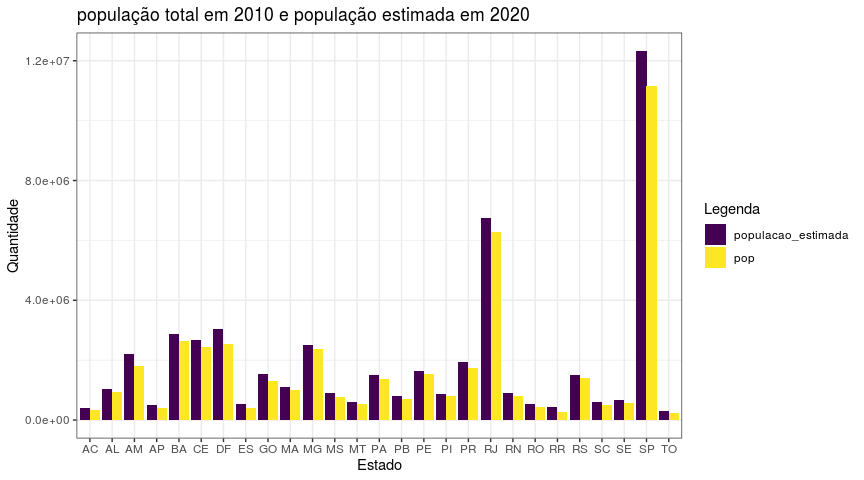
Ao olharmos brevemente para o gráfico, percebemos que a população estimada é maior em todos os Estados, ou seja, estima-se que a população de todos os Estados cresceu ao longos desses 10 anos. Destaque para o Estado de São Paulo que aparentemente é o que mais cresceu.
Para fechar o universo tidyverse, vamos falar por último (mas não
menos importante) do tibble e do purrr.
tibble
Um tibble se descreve como uma repaginação moderna dos dados.
Basicamente um tibble é um tipo de data.frame com caracteríscas
diferentes, pois não mudam os nomes ou tipos de variáveis e não fazem
correspondência parcial. Isso força você a enfrentar os problemas mais
cedos. Vamos então a um exemplo de uso.
notas <- c("André" = 6, "Larissa" = 9, "Mariana" = 8, "Tobias" = 3)
notas <- notas %>%
enframe(name = "aluno", value = "nota") #enframe converte vetores nomeados ou listas em quadros de dados de uma ou duas colunas
notas
## # A tibble: 4 x 2
## aluno nota
## <chr> <dbl>
## 1 André 6
## 2 Larissa 9
## 3 Mariana 8
## 4 Tobias 3
Quando rodamos notas no console, podemos ver que aparece uma mensagem
do tipo “# A tibble: 4 x 2”, ou seja, um tibble com comprimento 4 por
- Quando importamos banco de dados utilizando o pacote
readr, na maioria das vezes ele já vem no formato de umtibble, inclusive os dados dados que usamos para esse post.
purrr
O pacote purr veio para aprimorar as ferramentas de programação
funcional. Ele nos fornece um conjunto de ferramentas para trabalharmos
com vetores e funções. Você pode conferir o cheat sheet clicando
aqui.
Umas das funções mais importantes do pacote é a map. map é uma
abstração de laços, permitindo que façamos iterações nos elementos de
uma lista e não em alguma variável auxiliar. Em outras palavras ele
aplica uma função em todo elemento de uma lista. Suponha que a gente
queira calcular a diferença média entre população estimada e a população
real. Uma ideia legal para evitar possíveis if´s e for´s seria a
utilização do map. Vamos criar um vetor com os Estados de nosso
interesse e uma função que ira calcular essa diferença. Então, a função
map vai simplismente aplicar a função de interesse (definida no
argumento) em cada elemento do nosso vetor.
lista_estados <- c("ES", "SP", "RJ")
dfm <- function(a){
dadosf <- filter(dados, uf == a)
mean(dadosf$populacao_estimada - dadosf$pop)
}
map(lista_estados, dfm)
## [[1]]
## [1] 7396.545
##
## [[2]]
## [1] 8287.934
##
## [[3]]
## [1] 15981.25
Este post foi feito para mostrar um pouco de cada pacote do tidyverse
e a aplicação de algumas funções em um tratamento simples de dois bancos
de dados. Após entender o objetivo de cada pacote, fica mais fácil saber
onde procurar determinadas funções que poderão te auxiliar em
determinada etapa de sua análise. Vale lembrar que iremos fazer um post
mais aprofundado sobre esses pacotes, então fiquem ligados.
