Código
library(readr)
library(forcats) # trabalhar com fatores
library(ggthemes) # temas para gráficos
library(dplyr) # transformar dados
library(ggplot2) # fazer gráficos lindos
library(stringr) # para trabalhar com textosPedro de Brito Neto
20 de novembro de 2020
Diante da recente pandemia do novo coronavirus, ressalta-se a importância da discussão de políticas públicas para diferentes os grupos raciais da população brasileira. Aqui no Espírito Santo, foi criado o Observatório Capixaba da COVID-19 nas Favelas (OCOFA), com pesquisadores de diversas áreas: epidemiologia, estatística, ciências sociais, serviço social, geografia, design e comunicação. Em especial, o objetivo deste observatório é produzir dados sobre os impactos sociais da pandemia nas favelas e na população negra.
Com essa temática em mente, nesse post vamos discutir como utilizar dados do Painel da Covid-19 do ES para mostrar como podemos utilizar essas informações para produzir algumas análises importantes nesse momento.
Primeiramente vamos carregar alguns pacotes que iremos utilizar:
Após carregar os pacotes, iremos carregar o conjunto de dados. Uma forma de ler diretamente essa informação no R pode ser feita da seguinte forma.
Outra maneira de obter a planilha, é baixando-a diretamente pelo site e importando do seu computador, substituindo o caminho https://bi.static.es.gov.br/covid19/MICRODADOS.csv pelo local onde o arquivo está salvo no seu computador. Agora podemos começar a organizar os dados para fazermos as análises. Para a análise desse post, iremos utilizar a base de dados utilizada obtida no dia 16/11/2020.
Como queremos fazer a análise baseada nos casos confirmados, podemos começar aplicando um filtro para selecionarmos apenas esses casos. Depois disso, usaremos o pacote forcats para mudarmos os níveis da variável “Sexo”. Em seguida, vamos criar uma nova variável chamada “Idade” que vai receber apenas os dois primeiros dígitos da varíavel “IdadeNaNotificacao”.
Com essas filtragens, já podemos começar a gerar alguns gráficos interessantes como, por exemplo, a quantidade de casos confirmados separados por faixa etária, a quantidade de casos confirmados separados por Raça/Cor e um histograma de frequência separado por sexo. Vale ressaltar, que para gerar o histograma é necessário mudarmos a varíavel “Idade” para o tipo numeric do R, utilizando a função as.numeric.
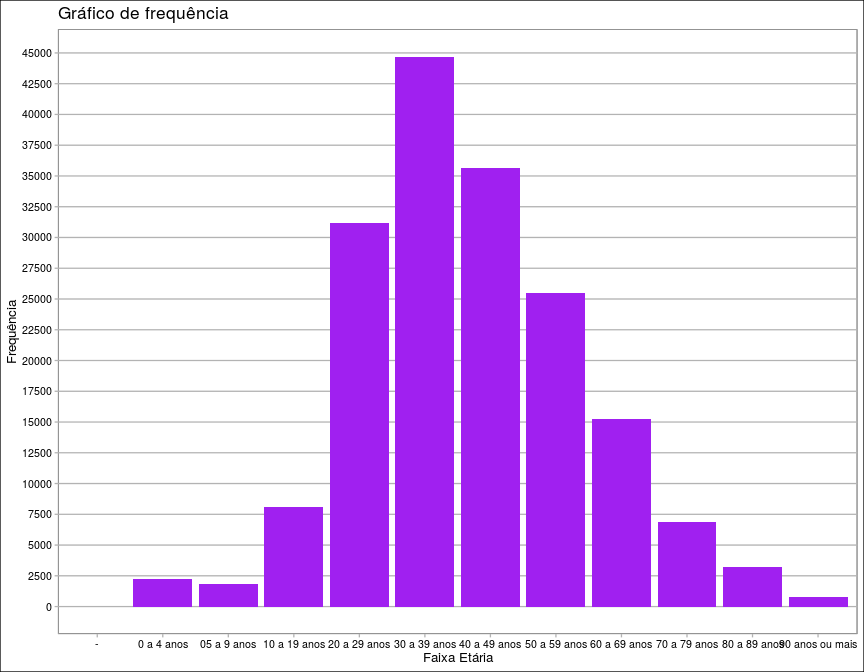
Neste gráfico podemos observar a frequência de casos confirmados de acordo com faixas etárias. Observamos que as faixas etárias de 30 a 39 anos, 20 a 29 anos e 40 a 49 anos foram as 3 faixas etárias com mais casos, respectivamente. Podemos notar também, que conforme a faixa etária vai aumentando, a frequência de casos confirmados vai diminuindo. Nota-se também que as faixas etárias entre 0 a 19 possui uma frequência menor, mas podemos observar um salto quando entramos no grupo de pessoas entre 20 e 29 anos.
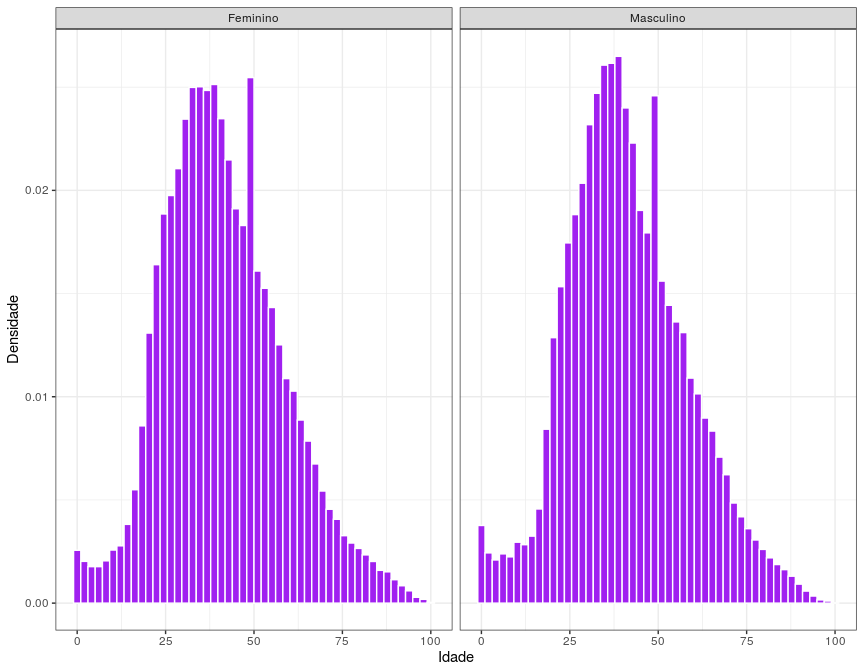
Para o próximo gráfico, aplicamos um filtro na variável sexo que removeu os dados classificados como “Ignorados”, para melhorar a visualização e a comparação entre os sexos “Masculino” e “Feminino”, já que existia uma quantidade pequena de ignorados.
Neste gráfico de histograma podemos comparar os sexos masculino e o feminino. Podemos notar que as duas distribuições de idade parece ser bem semelhantes entre os dois grupos.
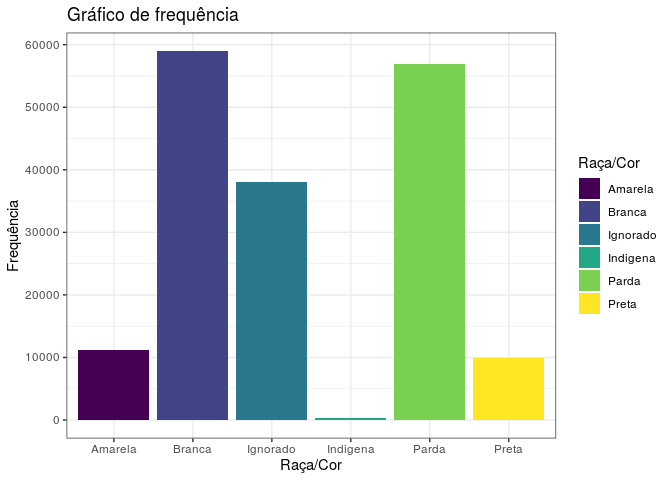
Ao observarmos esse gráfico, observamos que pessoas brancas e pardas tem uma presença muito grande na base de dados e possuem uma frequência bem proxima uma da outra. Nota-se também que existe uma grande quantidade de pessoas que tiveram sua raça/cor não preenchida e considerada então como “Ignorada”. A raça/cor indígena é a que possui a menor frequência dentre as presentes.
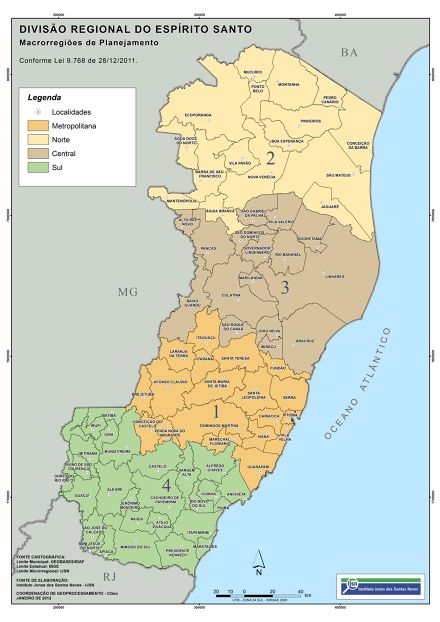
Uma análise interessante a ser feita é considerarmos os casos nas diferentes regiões do Espírito Santo. Seguimos a divisão feita pelo Governo do Estado que pode ser encontrada clicando aqui, onde o Estado foi dividido nas seguintes regiões: Metropolitana, Central, Norte e Sul. Esta divisão pode ser vista na figura a seguir.
Usamos uma função para atribuirmos cada cidade a sua devida região.
classifica_regiao <- function(Municipio) {
reg_metropolitana <- c(
"VITORIA",
"DOMINGOS MARTINS",
"SANTA LEOPOLDINA",
"MARECHAL FLORIANO",
"FUNDAO",
"SERRA",
"VILA VELHA",
"CARIACICA",
"VIANA",
"GUARAPARI",
"SANTA LEOPOLDINA",
"DOMIGOS MARTINS",
"MARCHAL FLORIANO",
"SANTA TERESA",
"SANTA MARIA DO JETIBA",
"ITAGUACU",
"LARANJA DA TERRA",
"ITARANA",
"AFONSO CLAUDIO",
"VENDA NOVA DO IMIGRANDE",
"BREJETUBA",
"CONCEICAO DO CASTELO"
)
reg_norte <- c(
"AGUIA BRANCA ",
"VENDA NOVA DO IMIGRANTE",
"SANTA MARIA DE JETIBA",
"MANTENOPOLIS",
"JAGUARE",
"SAO MATEUS",
"NOVA VENECIA",
"BARRA DE SAO FRANCISCO",
"VILA PAVAO",
"BOA ESPERANCA",
"AGUA DOCE DO NORTE",
"CONCEICAO DA BARRA",
"PINHEIROS",
"PEDRO CANARIO",
"MONTANHA",
"MUCURICI",
"PONTO BELO",
"ECOPORANGA"
)
reg_central <- c(
"ARACRUZ",
"IBIRACU",
"JOAO NEIVA",
"SAO ROQUE DO CANAA",
"BAIXO GUANDU",
"COLATINA",
"MARILANDIA",
"LINHARES",
"RIO BANANAL",
"PANCAS",
"SOORETAMA",
"GOVERNADOR LINDENBERG",
"SAO DOMINGOS DO NORTE",
"ALTO RIO NOVO",
"SAO GABRIEL DA PALHA",
"VILA VALERIO"
)
reg_sul <- c(
"PRESIDENTE KENNEDY",
"MUQUI",
"MARATAIZES",
"ITAPEMIRIM",
"MIMOSO DO SUL",
"APIACA",
"BOM JESUS DO NORTE",
"SAO JOSE DO CALCADO",
"MUQI",
"ATILIO VIVACQUA",
"PIUMA ",
"RIO NOVO DO SUL",
"CACHOEIRO DE ITAPEMIRIM",
"JERONIMO MONTEIRO",
"ALEGRE",
"GUACUI",
"DORES DO RIO PRETO",
"DIVINO DE SAO LOURENCO",
"IBITIRAMA",
"IUNA",
"IBATIBA",
"IRUPI",
"MUNIZ FREIRE",
"CASTELO",
"VARGEM ALTA",
"ALFREDO CHAVES",
"ANCHIETA",
"ICONHA",
"RIO NOVO DO SUL",
"PIUMA",
"ICONHA"
)
regiao <-
ifelse(any(Municipio == reg_metropolitana),
"Região Metropolitana",
ifelse(any(Municipio == reg_norte),
"Região Norte",
ifelse(any(Municipio == reg_central),
"Região Central",
ifelse(any(Municipio == reg_sul),
"Região Sul", "Outro Estado"
)
)
)
)
return(regiao)
}
dados$regiao <- sapply(dados$Municipio, classifica_regiao)Agora conseguimos observar construir um gráfico para observar a quantidade de casos por região:
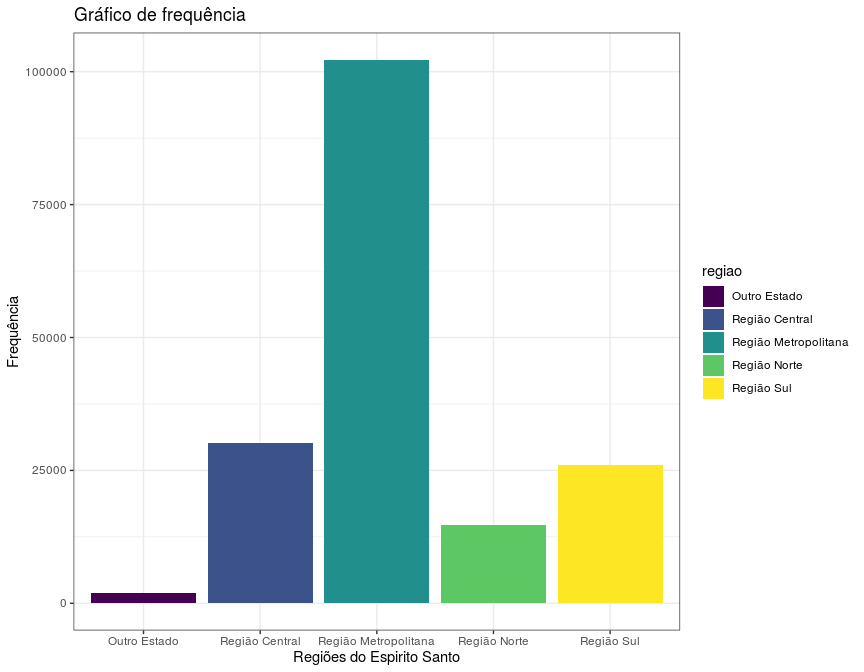
Podemos notar o quanto a região metropolitana se destaca em relação às outras, pois possui mais casos confirmados do que as outras 4 regiões juntas. Porém, deve-se ressaltar que tal região essa região é a mais populosa do Estado. A região norte, considerando as observações somente do Espírito Santo, foi a que menos registrou casos. Uma análise interessante que poderia ser feita, porém que não adicionaremos nesse post para não nos alongarmos, é comparar o número de confirmados pela população da região, o que nos daria um índice de incidência da doença. Isso ajudaria a entender melhor a diferença entre as regiões, porém isso não será analisado aqui nesse post.
Agora, iremos analisar em cada uma dessas regiões, os casos confirmados separados por sexo e raça/cor. Para essa análise, retiramos as observações de outros Estados. Utilizamos a função facet_wrap para separar os gráficos por região.
dados %>%
filter(regiao != "Outro Estado") %>%
ggplot() +
geom_bar(aes(RacaCor, fill = Sexo), position = "dodge") +
labs(y = "Frequência", x = "Raça/Cor") +
facet_wrap(~regiao, ncol = 2, scales = "free") +
theme_bw() +
scale_fill_viridis_d() +
theme(axis.text.x = element_text(angle = 45, vjust = 0.5))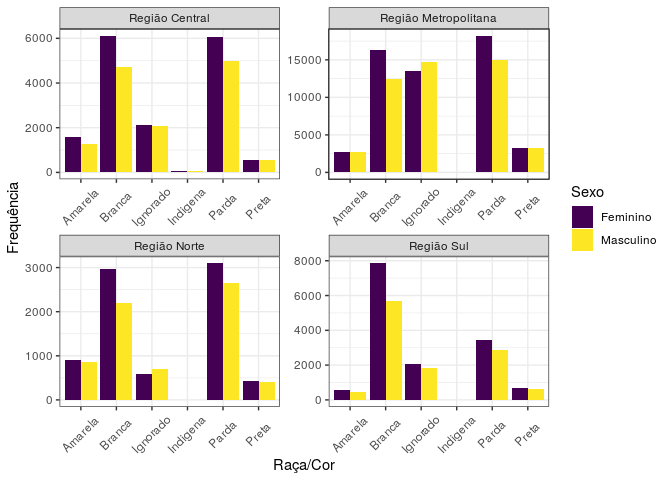
De forma geral, ao olharmos para a variável sexo, o número de casos para o sexo feminino é maior em relação ao sexo masculino em quase todas as situações. Se olharmos agora para a variável raça/cor, é visível que as classificações branca e parda têm uma frequência muito maior em relação às outras, enquanto que a raça indígena, apesar de presente nas 4 regiões, tem uma frequência maior na região central.
Podemos observar também a distribuição dos casos separados por sexo, ao longo do tempo.
dados <- dados %>%
mutate(DataNotificacao = as.Date(DataDiagnostico)) %>%
filter(DataNotificacao > as.Date("2020-01-01")) # retirando erro da base
casos_t <- dados %>%
group_by(Sexo, DataNotificacao) %>%
summarise(n_casos = n()) %>%
tidyr::complete(
tidyr::nesting(Sexo),
DataNotificacao = seq.Date(min(dados$DataNotificacao),
max(dados$DataNotificacao),
by = "day"
),
fill = list(n_casos = 0)
) %>%
mutate(casos_acumulados = cumsum(n_casos))
ggplot(casos_t) +
theme_minimal() +
aes(x = DataNotificacao, y = casos_acumulados, color = Sexo) +
geom_line() +
scale_color_viridis_d() +
labs(
y = "Número de casos acumulados",
x = "Data Diagnóstico",
title = "Casos acumulados por sexo"
)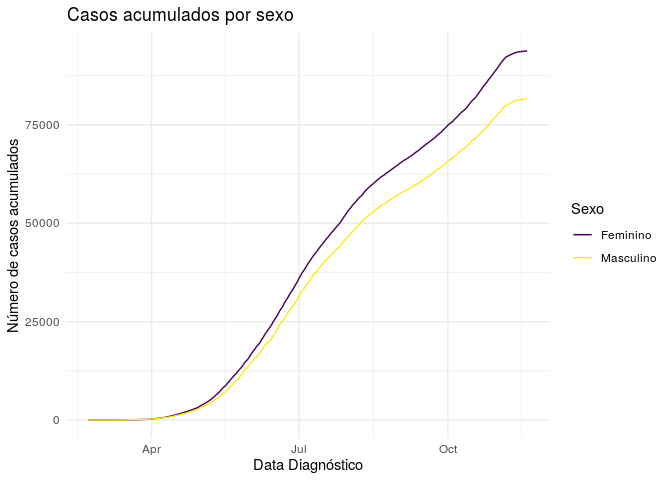
Observando o gráfico de casos acumulados separado por sexo, podemos ver de forma mais clara o que foi apontado anteriormente. O sexo femino tem uma frequência consideravelmente maior em relação ao sexo masculino, o que pode ser obervador por todo o intervalo de tempo analisado.
Por último, uma última análise interessante a ser feita, é calcular a taxa de letalidade da COVID-19. Para fazer isso precisamos calcular esse índice, podendo separar essa informação considerando algumas variáveis de interesse.
Para fazer essa tarefa criamos um data.frame chamado casos agrupando as variáveis “RacaCor” e “Sexo”, fazendo a contagem de casos confirmados. Depois, criamos um outro data.frame com as mesmas variáveis, porém esse objeto vai contar o número de mortes pela COVID-19. Por último, utilizamos a função inner.join para gerar o data.frame chamado “letalidade”, onde calculamos a taxa de letalidade da COVID-19. Vale ressaltar que a taxa de letalidade é a proporção entre o número de óbitos por uma doença em relação ao número de indivíduos acometidos por essa doença.
casos <- dados %>%
group_by(RacaCor, Sexo) %>%
summarise(n_casos = n())
mortes <- dados %>%
filter(Evolucao == "Óbito pelo COVID-19") %>%
group_by(RacaCor, Sexo) %>%
summarise(n_mortes = n()) %>%
tidyr::complete(RacaCor, Sexo,
fill = list(n_mortes = 0)
)
letalidade <- inner_join(casos, mortes) %>%
mutate(taxa_l = ifelse(n_casos == 0, 0,
round(100 * n_mortes / n_casos, 1)
))Por último podemos fazer o gráfico dessa informação:
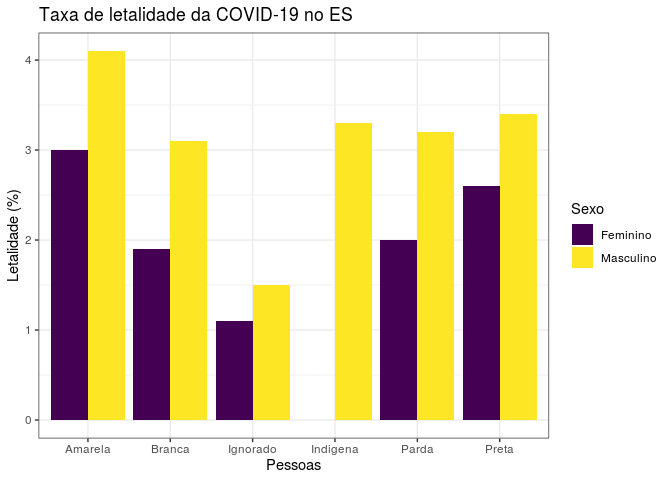
Ao observamos o gráfico, notamos que a taxa de letalidade do sexo masculino é sempre maior em relação ao sexo feminino em todas as raças. É possível notar também que para raça “Indígena” só observamos valores para o sexo “Masculino”. As maiores taxas para ambos os sexos foi observada na raça “Amarela”.
Existem diversas outras variáveis no banco de dados além daquelas que utilizamos aqui. A ideia desse post foi apenas ilustrar como utilizar algumas informações e como fazer alguns gráficos descritivos. Outras ideias de análise desse banco de dados também podem ser vistas no site do OCOFA.