Como usar o R para analisar dados da Covid-19 no Espírito Santo
Diante da recente pandemia do novo coronavirus, ressalta-se a importância da discussão de políticas públicas para diferentes os grupos raciais da população brasileira. Aqui no Espírito Santo, foi criado o Observatório Capixaba da COVID-19 nas Favelas (OCOFA), com pesquisadores de diversas áreas: epidemiologia, estatística, ciências sociais, serviço social, geografia, design e comunicação. Em especial, o objetivo deste observatório é produzir dados sobre os impactos sociais da pandemia nas favelas e na população negra.
Com essa temática em mente, nesse post vamos discutir como utilizar dados do Painel da Covid-19 do ES para mostrar como podemos utilizar essas informações para produzir algumas análises importantes nesse momento.
Primeiramente vamos carregar alguns pacotes que iremos utilizar:
library(readr)
library(forcats) # trabalhar com fatores
library(ggthemes) # temas para gráficos
library(dplyr) # transformar dados
library(ggplot2) # fazer gráficos lindos
library(stringr) # para trabalhar com textos
Após carregar os pacotes, iremos carregar o conjunto de dados. Uma forma de ler diretamente essa informação no R pode ser feita da seguinte forma.
dados <- read.csv("https://bi.static.es.gov.br/covid19/MICRODADOS.csv",
sep = ";",
fileEncoding = "ISO-8859-1")
dados <- read.csv("MICRODADOS.csv",
sep = ";")
Outra maneira de obter a planilha, é baixando-a diretamente pelo site e
importando do seu computador, substituindo o caminho
https://bi.static.es.gov.br/covid19/MICRODADOS.csv pelo local onde o
arquivo está salvo no seu computador. Agora podemos começar a organizar
os dados para fazermos as análises. Para a análise desse post, iremos
utilizar a base de dados utilizada obtida no dia 16/11/2020.
Preparando a base de dados
Como queremos fazer a análise baseada nos casos confirmados, podemos
começar aplicando um filtro para selecionarmos apenas esses casos.
Depois disso, usaremos o pacote forcats para mudarmos os níveis da
variável “Sexo”. Em seguida, vamos criar uma nova variável chamada
“Idade” que vai receber apenas os dois primeiros dígitos da varíavel
“IdadeNaNotificacao”.
dados <- filter(dados, Classificacao == "Confirmados")
dados <- dados %>%
mutate(Sexo = fct_recode(Sexo,
Masculino ="M",
Feminino = "F",
Ignorados = "I"))
dados <- dados %>%
mutate(Idade = str_sub(IdadeNaDataNotificacao, end = 2))
Com essas filtragens, já podemos começar a gerar alguns gráficos
interessantes como, por exemplo, a quantidade de casos confirmados
separados por faixa etária, a quantidade de casos confirmados separados
por Raça/Cor e um histograma de frequência separado por sexo. Vale
ressaltar, que para gerar o histograma é necessário mudarmos a varíavel
“Idade” para o tipo numeric do R, utilizando a função
as.numeric.
Analisando a variável idade
ggplot(dados) +
geom_bar(aes(x = FaixaEtaria), fill = "purple") +
labs(y = "Frequência",
x = "Faixa Etária",
title = "Gráfico de frequência ") +
scale_y_continuous(breaks = seq(0, 100000, 2500)) +
theme_calc()
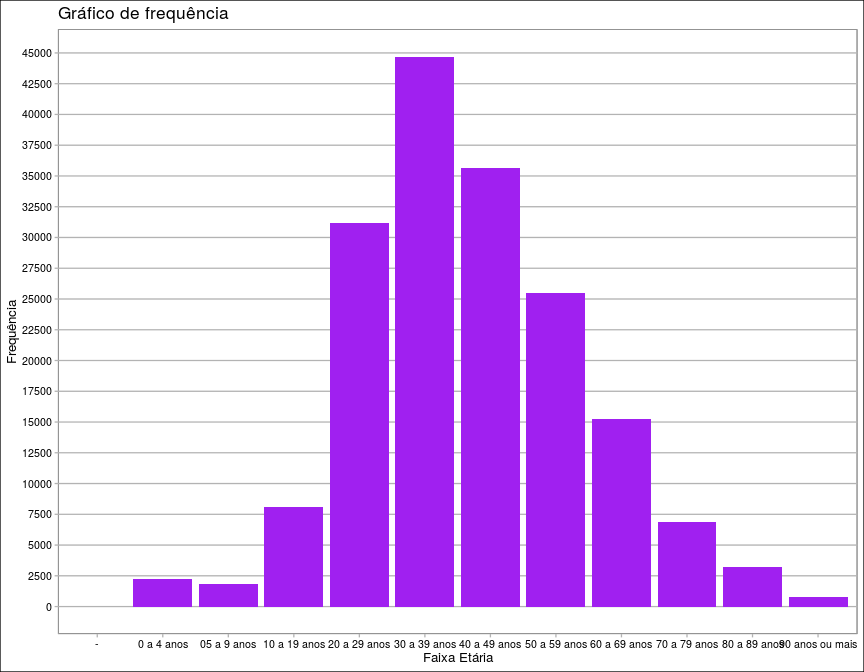
Neste gráfico podemos observar a frequência de casos confirmados de acordo com faixas etárias. Observamos que as faixas etárias de 30 a 39 anos, 20 a 29 anos e 40 a 49 anos foram as 3 faixas etárias com mais casos, respectivamente. Podemos notar também, que conforme a faixa etária vai aumentando, a frequência de casos confirmados vai diminuindo. Nota-se também que as faixas etárias entre 0 a 19 possui uma frequência menor, mas podemos observar um salto quando entramos no grupo de pessoas entre 20 e 29 anos.
Para o próximo gráfico, aplicamos um filtro na variável sexo que removeu os dados classificados como “Ignorados”, para melhorar a visualização e a comparação entre os sexos “Masculino” e “Feminino”, já que existia uma quantidade pequena de ignorados.
# Retirando as observações com Sexo == "Ignorados"
dados <- filter(dados, Sexo != "Ignorados")
Neste gráfico de histograma podemos comparar os sexos masculino e o feminino. Podemos notar que as duas distribuições de idade parece ser bem semelhantes entre os dois grupos.
dados <- dados %>%
mutate(Idade = as.numeric(Idade))
ggplot(dados, aes(x = Idade, y = ..density..)) +
geom_histogram(color = "white", bins = 50, fill = 'purple') +
facet_wrap(~ Sexo) +
theme_bw() +
ylab("Densidade") +
scale_fill_viridis_d()

Analisando por raça/cor
ggplot(dados) +
geom_bar(aes(x = RacaCor, fill = RacaCor)) +
scale_y_continuous(breaks = seq(0, 200000, 10000)) +
labs(y = "Frequência",
x = "Raça/Cor",
title = "Gráfico de frequência") +
theme_bw() +
scale_fill_viridis_d(name = "Raça/Cor")
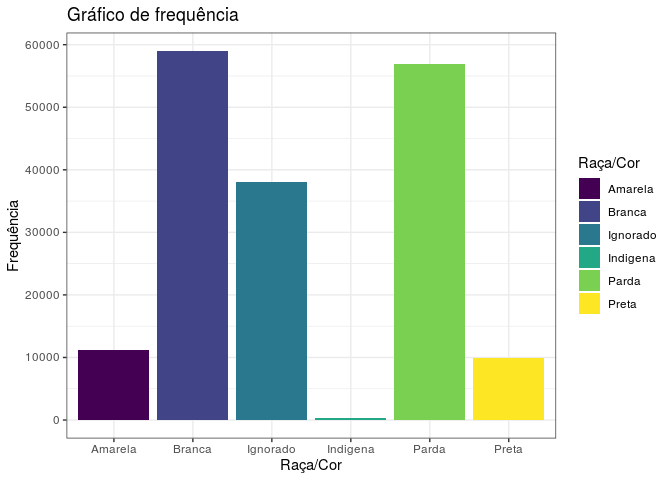
Ao observarmos esse gráfico, observamos que pessoas brancas e pardas tem uma presença muito grande na base de dados e possuem uma frequência bem proxima uma da outra. Nota-se também que existe uma grande quantidade de pessoas que tiveram sua raça/cor não preenchida e considerada então como “Ignorada”. A raça/cor indígena é a que possui a menor frequência dentre as presentes.
Análise por região
Uma análise interessante a ser feita é considerarmos os casos nas diferentes regiões do Espírito Santo. Seguimos a divisão feita pelo Governo do Estado que pode ser encontrada clicando aqui, onde o Estado foi dividido nas seguintes regiões: Metropolitana, Central, Norte e Sul. Esta divisão pode ser vista na figura a seguir.
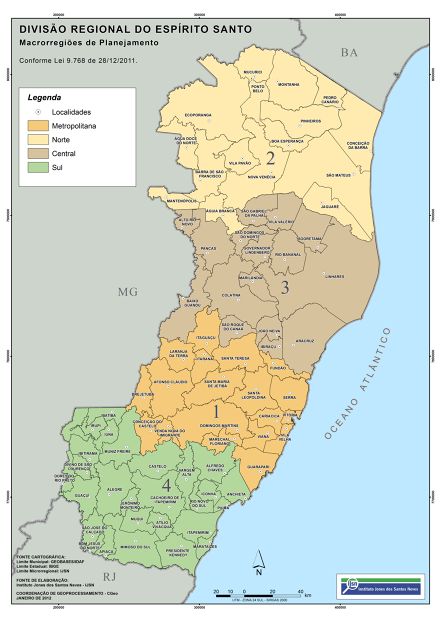
Usamos uma função para atribuirmos cada cidade a sua devida região.
classifica_regiao <- function(Municipio){
reg_metropolitana <- c(
'VITORIA',
"DOMINGOS MARTINS" ,
"SANTA LEOPOLDINA" ,
"MARECHAL FLORIANO" ,
'FUNDAO' ,
'SERRA' ,
'VILA VELHA' ,
'CARIACICA' ,
'VIANA' ,
'GUARAPARI' ,
"SANTA LEOPOLDINA" ,
"DOMIGOS MARTINS" ,
"MARCHAL FLORIANO" ,
"SANTA TERESA" ,
"SANTA MARIA DO JETIBA" ,
"ITAGUACU" ,
"LARANJA DA TERRA" ,
"ITARANA" ,
"AFONSO CLAUDIO" ,
"VENDA NOVA DO IMIGRANDE" ,
"BREJETUBA" ,
"CONCEICAO DO CASTELO"
)
reg_norte <- c(
"AGUIA BRANCA " ,
"VENDA NOVA DO IMIGRANTE" ,
"SANTA MARIA DE JETIBA" ,
"MANTENOPOLIS" ,
"JAGUARE" ,
"SAO MATEUS" ,
"NOVA VENECIA" ,
"BARRA DE SAO FRANCISCO" ,
"VILA PAVAO" ,
"BOA ESPERANCA" ,
"AGUA DOCE DO NORTE" ,
"CONCEICAO DA BARRA" ,
"PINHEIROS" ,
"PEDRO CANARIO" ,
"MONTANHA" ,
"MUCURICI" ,
"PONTO BELO" ,
"ECOPORANGA"
)
reg_central <- c(
"ARACRUZ" ,
"IBIRACU" ,
"JOAO NEIVA" ,
"SAO ROQUE DO CANAA" ,
"BAIXO GUANDU" ,
"COLATINA" ,
"MARILANDIA" ,
"LINHARES" ,
"RIO BANANAL" ,
"PANCAS" ,
"SOORETAMA" ,
"GOVERNADOR LINDENBERG" ,
"SAO DOMINGOS DO NORTE" ,
"ALTO RIO NOVO" ,
"SAO GABRIEL DA PALHA" ,
"VILA VALERIO"
)
reg_sul <- c(
'PRESIDENTE KENNEDY' ,
"MUQUI" ,
'MARATAIZES' ,
'ITAPEMIRIM' ,
'MIMOSO DO SUL' ,
'APIACA' ,
'BOM JESUS DO NORTE' ,
'SAO JOSE DO CALCADO' ,
"MUQI" ,
"ATILIO VIVACQUA" ,
"PIUMA " ,
"RIO NOVO DO SUL" ,
"CACHOEIRO DE ITAPEMIRIM" ,
"JERONIMO MONTEIRO" ,
"ALEGRE" ,
"GUACUI" ,
"DORES DO RIO PRETO" ,
"DIVINO DE SAO LOURENCO" ,
"IBITIRAMA" ,
"IUNA" ,
"IBATIBA" ,
"IRUPI" ,
"MUNIZ FREIRE" ,
"CASTELO" ,
"VARGEM ALTA" ,
"ALFREDO CHAVES" ,
"ANCHIETA" ,
"ICONHA" ,
"RIO NOVO DO SUL" ,
"PIUMA" ,
"ICONHA"
)
regiao <-
ifelse(any(Municipio == reg_metropolitana),
"Região Metropolitana",
ifelse(any(Municipio == reg_norte),
"Região Norte",
ifelse(any(Municipio == reg_central),
"Região Central",
ifelse(any(Municipio == reg_sul),
"Região Sul", "Outro Estado")
)
)
)
return(regiao)
}
dados$regiao <- sapply(dados$Municipio, classifica_regiao)
Agora conseguimos observar construir um gráfico para observar a quantidade de casos por região:
ggplot(dados) + geom_bar(aes(x = regiao, fill = regiao)) +
#scale_y_continuous(breaks = seq(0, 1000000, 10000)) +
labs(y = "Frequência",
x = "Regiões do Espirito Santo",
title = "Gráfico de frequência") +
theme_bw() +
scale_fill_viridis_d() +
facet_grid(scales = "free")
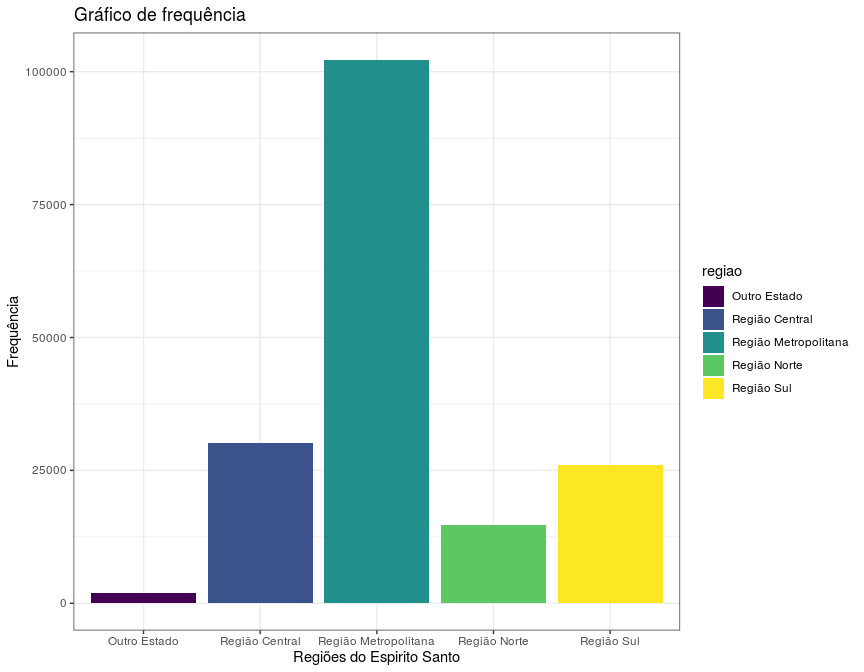
Podemos notar o quanto a região metropolitana se destaca em relação às outras, pois possui mais casos confirmados do que as outras 4 regiões juntas. Porém, deve-se ressaltar que tal região essa região é a mais populosa do Estado. A região norte, considerando as observações somente do Espírito Santo, foi a que menos registrou casos. Uma análise interessante que poderia ser feita, porém que não adicionaremos nesse post para não nos alongarmos, é comparar o número de confirmados pela população da região, o que nos daria um índice de incidência da doença. Isso ajudaria a entender melhor a diferença entre as regiões, porém isso não será analisado aqui nesse post.
Agora, iremos analisar em cada uma dessas regiões, os casos confirmados
separados por sexo e raça/cor. Para essa análise, retiramos as
observações de outros Estados. Utilizamos a função facet_wrap para
separar os gráficos por região.
dados %>%
filter(regiao != "Outro Estado") %>%
ggplot() +
geom_bar(aes(RacaCor, fill = Sexo), position = "dodge") +
labs(y = "Frequência", x = "Raça/Cor") +
facet_wrap(~ regiao, ncol = 2, scales = "free") +
theme_bw() +
scale_fill_viridis_d() +
theme(axis.text.x = element_text(angle = 45, vjust = 0.5))
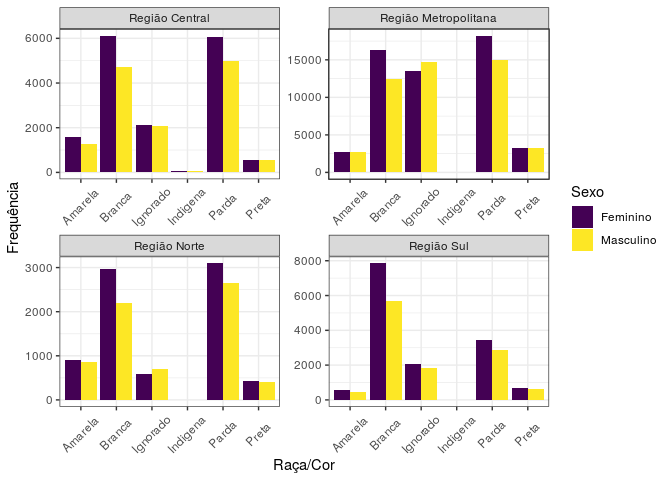
De forma geral, ao olharmos para a variável sexo, o número de casos para o sexo feminino é maior em relação ao sexo masculino em quase todas as situações. Se olharmos agora para a variável raça/cor, é visível que as classificações branca e parda têm uma frequência muito maior em relação às outras, enquanto que a raça indígena, apesar de presente nas 4 regiões, tem uma frequência maior na região central.
Podemos observar também a distribuição dos casos separados por sexo, ao longo do tempo.
dados <- dados %>%
mutate(DataNotificacao = as.Date(DataDiagnostico)) %>%
filter(DataNotificacao > as.Date("2020-01-01")) # retirando erro da base
casos_t <- dados %>%
group_by(Sexo, DataNotificacao) %>%
summarise(n_casos = n()) %>%
tidyr::complete(
tidyr::nesting(Sexo),
DataNotificacao = seq.Date(min(dados$DataNotificacao),
max(dados$DataNotificacao),
by = "day"),
fill = list(n_casos = 0)
) %>%
mutate(casos_acumulados = cumsum(n_casos))
ggplot(casos_t) +
theme_minimal() +
aes(x = DataNotificacao, y = casos_acumulados, color = Sexo) +
geom_line() +
scale_color_viridis_d() +
labs(y = "Número de casos acumulados",
x = "Data Diagnóstico",
title = "Casos acumulados por sexo")
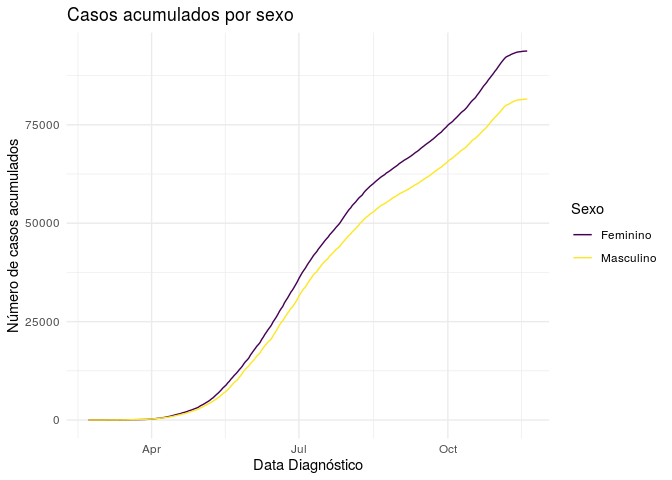
Observando o gráfico de casos acumulados separado por sexo, podemos ver de forma mais clara o que foi apontado anteriormente. O sexo femino tem uma frequência consideravelmente maior em relação ao sexo masculino, o que pode ser obervador por todo o intervalo de tempo analisado.
Por último, uma última análise interessante a ser feita, é calcular a taxa de letalidade da COVID-19. Para fazer isso precisamos calcular esse índice, podendo separar essa informação considerando algumas variáveis de interesse.
Para fazer essa tarefa criamos um data.frame chamado casos agrupando
as variáveis “RacaCor” e “Sexo”, fazendo a contagem de casos
confirmados. Depois, criamos um outro data.frame com as mesmas
variáveis, porém esse objeto vai contar o número de mortes pela
COVID-19. Por último, utilizamos a função inner.join para gerar o
data.frame chamado “letalidade”, onde calculamos a taxa de letalidade
da COVID-19. Vale ressaltar que a taxa de letalidade é a proporção entre
o número de óbitos por uma doença em relação ao número de indivíduos
acometidos por essa doença.
casos <- dados %>%
group_by(RacaCor, Sexo) %>%
summarise(n_casos = n())
mortes <- dados %>%
filter(Evolucao == "Óbito pelo COVID-19") %>%
group_by(RacaCor, Sexo) %>%
summarise(n_mortes = n()) %>%
tidyr::complete(RacaCor, Sexo,
fill = list( n_mortes = 0))
letalidade <- inner_join(casos, mortes) %>%
mutate(taxa_l = ifelse(n_casos == 0, 0,
round(100 * n_mortes / n_casos, 1)))
Por último podemos fazer o gráfico dessa informação:
ggplot(letalidade) +
geom_col(aes(x = RacaCor, y = taxa_l, fill = Sexo), position = "dodge") +
labs(x = "Pessoas",
y = "Letalidade (%)",
title = "Taxa de letalidade da COVID-19 no ES") +
scale_fill_viridis_d() +
theme_bw()
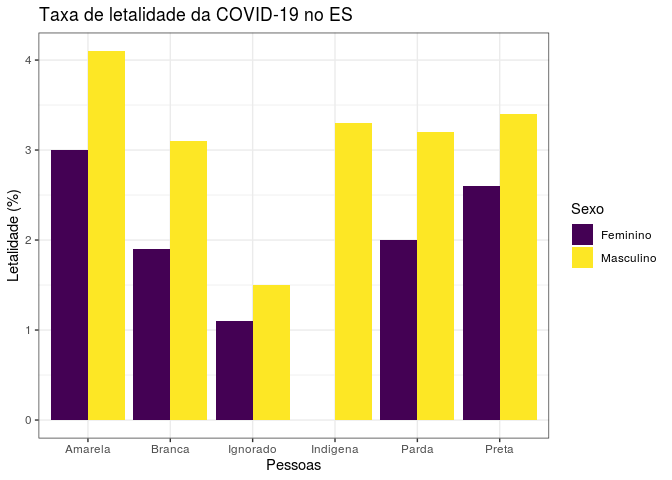
Ao observamos o gráfico, notamos que a taxa de letalidade do sexo masculino é sempre maior em relação ao sexo feminino em todas as raças. É possível notar também que para raça “Indígena” só observamos valores para o sexo “Masculino”. As maiores taxas para ambos os sexos foi observada na raça “Amarela”.
Existem diversas outras variáveis no banco de dados além daquelas que utilizamos aqui. A ideia desse post foi apenas ilustrar como utilizar algumas informações e como fazer alguns gráficos descritivos. Outras ideias de análise desse banco de dados também podem ser vistas no site do OCOFA.
